Abstracto
La resistencia a los antimicrobianos (RAM) representa una amenaza global para la salud pública. Si bien el uso excesivo de antibióticos es un factor principal, la evidencia emergente sugiere que los medicamentos no antibióticos (NAM) también pueden contribuir. Esta preocupación es particularmente relevante en los centros residenciales de atención a personas mayores (RACF), donde se usan con frecuencia tanto NAM como antibióticos. Investigamos si nueve NAM de uso común en RACF, incluidos ibuprofeno, diclofenaco, acetaminofén, furosemida, metformina, atorvastatina, tramadol, temazepam y pseudoefedrina en concentraciones relevantes para el intestino, mejoran la mutagénesis inducida por ciprofloxacino en Escherichia coli . Nuestros hallazgos mostraron que el ibuprofeno y el acetaminofén aumentaron significativamente la frecuencia de mutación y confirieron resistencia de alto nivel a la ciprofloxacina. La secuenciación del genoma completo identificó mutaciones en GyrA, MarR y AcrR, y las dos últimas se correlacionaron con la sobreexpresión de la bomba de eflujo de fármacos AcrAB-TolC. La coexposición a dos NAM aumentó aún más las tasas de mutación y los niveles de resistencia a la ciprofloxacina. Este estudio subrayó el papel poco conocido de los NAM en el aumento de la RAM y destacó la necesidad de reevaluar los riesgos de la polifarmacia en centros de atención a personas mayores.
El desarrollo y la propagación de organismos resistentes a los antimicrobianos y genes de resistencia representan una de las mayores amenazas que enfrenta la salud pública 1 . Esto se ejemplifica con los informes actuales que estiman que 4,95 millones de muertes estuvieron asociadas con la resistencia a los antimicrobianos en 2019 2 . Es bien sabido que el amplificador prevalente detrás del desarrollo de la resistencia a los antimicrobianos (RAM) es el uso excesivo de antibióticos en entornos clínicos y agrícolas 3 , 4 . Sin embargo, además de los antibióticos, el uso de medicamentos no antibióticos (NAMs) como diuréticos, estatinas, inhibidores de la bomba de protones, entre otros, también han demostrado contribuir al aumento de la RAM 5 , 6 .
Un informe reciente que evaluó las propiedades antimicrobianas de los NAM reveló que más de 200 medicamentos de uso común poseían efectos similares a los antibióticos en las bacterias intestinales 7 . Actualmente, el mercado farmacéutico mundial está compuesto principalmente por medicamentos no antibióticos, que ocupan el 95% del mercado 8 . El mercado farmacéutico también sigue creciendo, experimentando un crecimiento significativo durante los últimos dos años, con ingresos mundiales que totalizaron 1,48 billones (de dólares estadounidenses) en 2022 9 . El crecimiento de esta industria no es sorprendente, ya que la población mundial está envejeciendo 10 , y esta población envejecida depende de un arsenal de medicamentos para controlar las enfermedades crónicas 11 , y algunos toman nueve o más medicamentos recetados por día, un fenómeno conocido como polifarmacia, y que está aumentando en todo el mundo 11 , 12 , 13 , 14 . Las personas mayores también representan una población con un alto consumo de antibióticos, que con frecuencia se usan en exceso en los centros de atención residencial para ancianos (RACF) para tratar infecciones de las vías urinarias y respiratorias 15 , 16 , 17 , 18 , 19 .
Los NAM pueden tener un efecto directo o indirecto en el desarrollo de la RAM. Algunos NAM muestran actividad antibacteriana directa, como las estatinas 20 . Otros, como ciertos fármacos antiinflamatorios no esteroideos (AINE), pueden promover el desarrollo de resistencia cruzada. Esto implica seleccionar mecanismos de resistencia que confieran simultáneamente resistencia a los antibióticos, siendo las bombas de eflujo de múltiples fármacos un excelente ejemplo 21 . Las bacterias también pueden adquirir mecanismos de resistencia adicionales al adoptar un plásmido que expresa factores de resistencia en un proceso denominado transformación. El diclofenaco es un ejemplo de un NAM que podría aumentar la eficiencia de la transformación con el consiguiente aumento de la resistencia adquirida 6 . A esto se suma el hecho de que algunos antibióticos como la ciprofloxacina, una fluoroquinolona, y fármacos no antibióticos como el tramadol, un opioide débil, no se degradan completamente en el cuerpo y terminan en las aguas residuales a través de la excreción de orina o heces 22 . Una vez allí, estos y los metabolitos de los antibióticos y de los fármacos no antibióticos ejercen su efecto combinado y crean un entorno perfecto para el desarrollo y la diseminación de la RAM.
Para evaluar la propensión de los NAM a promover el desarrollo de RAM, investigamos nueve medicamentos; acetaminofén, ibuprofeno, diclofenaco, furosemida, atorvastatina cálcica, metformina, pseudoefedrina, temazepam y tramadol, que son comúnmente utilizados por personas mayores 23 , 24 . Como estos NAM se coadministran frecuentemente con antibióticos, se determinaron las frecuencias de mutación en presencia de ciprofloxacino. El ciprofloxacino no solo es un inductor conocido de mutaciones, sino que también se usa en el tratamiento de infecciones del tracto urinario (ITU), que son una de las infecciones bacterianas más comunes 25 , 26 . El uso frecuente de ciprofloxacino en entornos RACF ha llevado a la aparición de resistencia al ciprofloxacino y Escherichia coli resistente al ciprofloxacino , lo que ha representado una amenaza para los residentes que viven en RACF 27 . Se determinó el efecto de los nueve NAM y las combinaciones de dos NAM en las frecuencias de mutación de dos aislados de E. coli : E. coli BW25113, un derivado de E. coli K-12, y E. coli 6146, aislado de una muestra fecal de un residente de una residencia de ancianos. La susceptibilidad antimicrobiana de los mutantes inducidos por NAM y los mecanismos subyacentes a la resistencia observada se evaluaron mediante secuenciación genómica completa. También se exploró en mayor profundidad el papel de las bombas de eflujo de fármacos, que pueden sobreexpresarse en respuesta al estrés y contribuir a la resistencia cruzada.
Resultados
La exposición a NAM aumenta la aptitud de E. coli en presencia de ciprofloxacino
Para evaluar la propensión de los no antibióticos de uso común a inducir resistencia bacteriana a los antibióticos, se eligieron seis NAM en función de su uso frecuente entre los ancianos en RACF 28 , 29 . En este estudio se utilizó ciprofloxacino por ser un inductor conocido de mutaciones 30 con un aumento de uropatógenos resistentes al ciprofloxacino que representan una amenaza para los residentes que viven en centros de atención para ancianos 27 , 31 , 32 , 33 . Las frecuencias de mutación se determinaron en E. coli BW25113 y E. coli 6146 (una cepa sensible a los antibióticos aislada de un residente jubilado). Se determinó la susceptibilidad antimicrobiana basal de las cepas de prueba, E. coli BW25113 y E. coli 6146, y se demostró que era casi idéntica y de tipo salvaje para los agentes probados (Tabla 1 ). Ninguno de los NAM evaluados en este estudio mostró actividad antimicrobiana contra estos aislados, incluso a la concentración más alta analizada (512 μg/mL). Para obtener un conjunto completo de datos de susceptibilidad a los antibióticos, también se incluyeron la eritromicina y la novobiocina, a las que se sabe que las cepas de E. coli son resistentes . 34
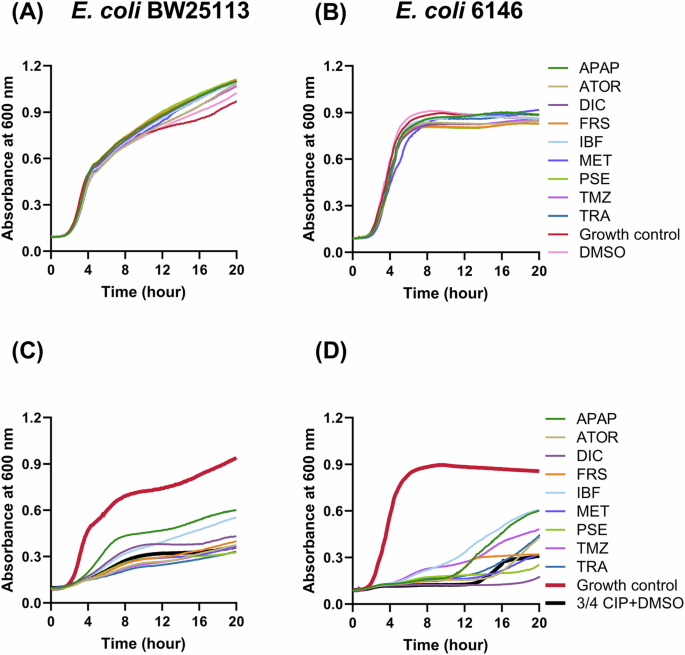
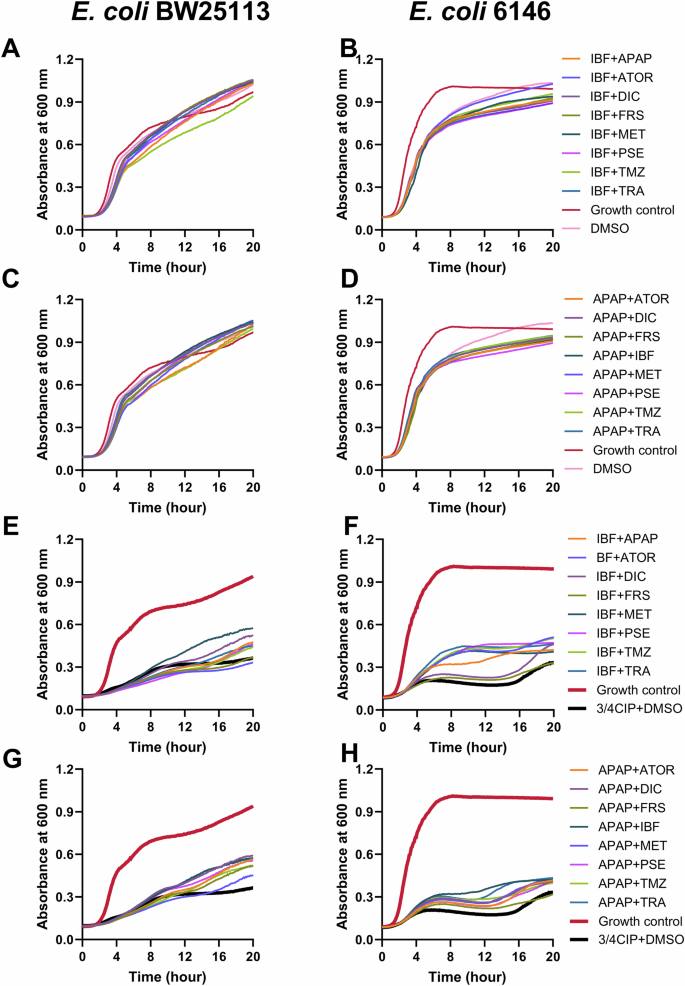
Para investigar si el crecimiento se vio afectado por la exposición a NAM, se analizó el crecimiento de E. coli BW25113 y E. coli 6146 en presencia de concentraciones subinhibitorias de ciprofloxacino y/o concentraciones intestinales de los NAM analizados en este estudio (Fig. 1 ) . Como se esperaba, en comparación con las células no expuestas, la adición de NAM por sí sola no afectó al crecimiento de las células de E. coli (Fig. 1A, B ). Sin embargo, la tasa de crecimiento y la densidad celular máxima se vieron drásticamente afectadas a 3/4 de la CMI de ciprofloxacino para las células de E. coli BW25113 y E. coli 6146. Se observó una reducción del crecimiento celular de 0,457 ± 0,009 h − 1 a 0,140 ± 0,006 h −1 y de 0,443 ± 0,006 h −1 a 0,007 ± 0,001 h −1 para E. coli BW25113 y E. coli 6146, respectivamente (Fig. 1C, D ). Además, el inicio de la tasa de crecimiento exponencial se retrasó para las células de E. coli 6146 en presencia de ciprofloxacino (Fig. 1D ). Mientras que en presencia de ¾ de la CMI de ciprofloxacino, diclofenaco, ibuprofeno y acetaminofén aumentaron las tasas de crecimiento de E. coli BW25113 a 0,201 ± 0,004, 0,179 ± 0,006 y 0,2823 ± 0,013 h −1 respectivamente (Fig. 1C ). Los mismos tres NAM también pudieron reducir la duración de la fase de latencia (Fig. 1D ). En conjunto, ciertos NAM aumentan la aptitud y la adaptabilidad de E. coli en condiciones de estrés, como el crecimiento en presencia de un antibiótico.
Perfiles de crecimiento de E. coli BW25113 y 6146 en presencia de ciprofloxacino subinhibitorio y NAM. A , B El efecto de los NAM individuales en el crecimiento de E. coli BW25113 y E. coli 6146. C , D El efecto de los NAM individuales en el crecimiento de E. coli BW25113 y E. coli 6146 en presencia de ¾ MIC de ciprofloxacino. El control de crecimiento representó células en ausencia de NAM y ciprofloxacino. APAP Acetaminofén, ATOR atorvastatina cálcica, CIP ciprofloxacino, DIC diclofenaco, FRS furosemida, IBF ibuprofeno, MET metformina, PSE pseudoefedrina, TMZ temazepam, TRA tramadol. Las curvas de crecimiento se realizaron cuatro veces en días diferentes con diferentes lotes de células. Aquí se muestran trazas representativas de triplicados técnicos, seleccionados en función de los resultados generales.
La exposición a NAM induce mutaciones dentro de E. coli
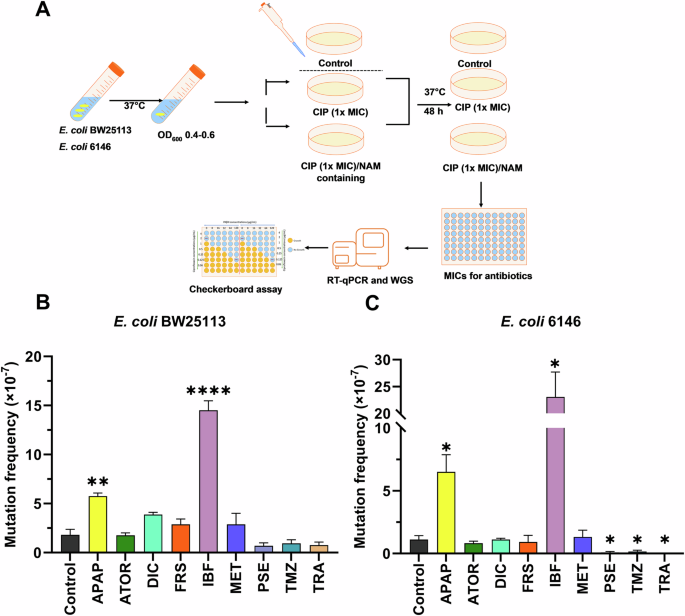
Para investigar el potencial de los NAM evaluados en este estudio para inducir el desarrollo de RAM por mutación, las células de E. coli BW25113 y 6146 se expusieron a NAM en las concentraciones intestinales proyectadas y ciprofloxacino a 1 x la CMI (0,015 µg/mL) y se cultivaron durante 48 horas (Fig. 2A ) 7 . Todos los ensayos se llevaron a cabo en placas suplementadas con 0,015 µg/ml de ciprofloxacino (1x CMI), ya que no se observaron colonias (mutantes) a concentraciones más altas de ciprofloxacino, incluidas 2x y 4x CMI.
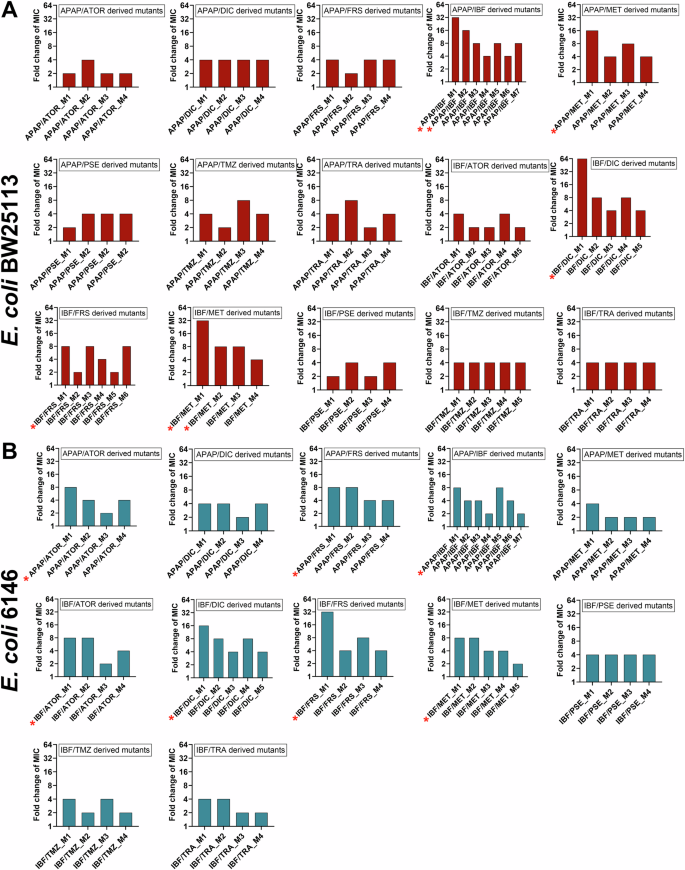
Un flujo de trabajo utilizado para determinar las frecuencias de mutación de las células E. coli BW25113 y 6146 expuestas a NAM y ciprofloxacina. B, C La frecuencia de mutación de E. coli BW25113 y E. coli 6146 se determinó tras la exposición a NAM a 216 μg/mL para acetaminofeno (APAP), 6,6 μg/mL para atorvastatina (ATOR), 15,4 μg/mL para diclofenaco (DIC), 13,2 μg/mL para furosemida (FRS), 99,8 μg/mL para ibuprofeno (IBF), 194,6 μg/mL para metformina (MET), 40 μg/mL para pseudoefedrina (PSE), 3,3 μg/mL para temazepam (TMZ) y 33,3 μg/mL para tramadol (TRA) y ciprofloxacino (CIP) (0,015 μg/mL). Las diferencias entre los mutantes tratados con ciprofloxacino/NAM y el grupo control (ciprofloxacino tratado con DMSO) se analizaron mediante pruebas t de muestras independientes y se corrigieron mediante el método de Bonferroni: * P < 0,05, ** P < 0,01, *** P < 0,001 y **** P < 0,0001. Los datos se expresan como media ± EEM de cuatro experimentos realizados en días diferentes con distintos lotes de células.
Se observaron frecuencias de mutación más altas para E. coli BW25113, con (1,45 ± 0,19) × 10 -6 y (5,75 ± 0,64) × 10 −7 células expuestas a ibuprofeno ( P < 0,0001) y acetaminofén ( p < 0,01), respectivamente, en comparación con la exposición a ciprofloxacino solo (Fig. 2 ). De manera similar, se observó un aumento significativo en la frecuencia de mutación para E. coli 6146 después de la exposición a acetaminofén (6,50 ± 2,73) × 10 −7 e ibuprofeno (2,30 ± 0,92) × 10 −6 . Cabe destacar que las frecuencias de mutación de pseudoefedrina, temazepam y tramadol mostraron una disminución significativa para E. coli 6146 en comparación con la exposición a ciprofloxacino solo. Las frecuencias de mutación de los demás NAM evaluados en este estudio no fueron significativamente diferentes de las del tratamiento con ciprofloxacino solo, aunque las frecuencias de mutación para E. coli BW25113 fueron mayores que para la cepa clínica cuando las células se expusieron a ciprofloxacino en combinación con diclofenaco, atorvastatina, furosemida y metformina (Fig. 2 ).
La exposición a NAM provoca una elevada resistencia a la ciprofloxacina en aislados seleccionados
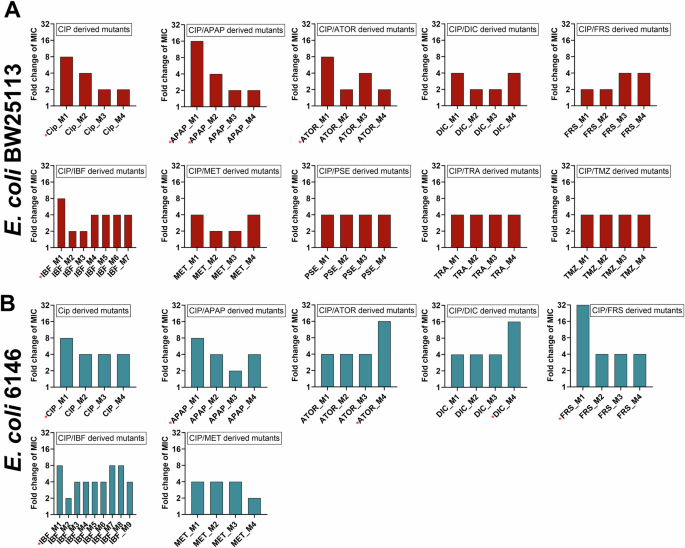
Para investigar los niveles de resistencia adquirida a la ciprofloxacina, se seleccionó un número representativo (√n + 1) de mutantes derivados de E. coli BW25113 y 6146 de cada placa de combinación de ciprofloxacina/NAM y se determinó la MIC de ciprofloxacina (Fig. 3 ) . Como se esperaba, las células de E. coli BW25113 expuestas a ciprofloxacina solo desarrollaron resistencia, lo que resultó en un aumento de las MIC de ciprofloxacina (aumento de 2 a 8 veces en la MIC de ciprofloxacina en comparación con E. coli WT ). También se observó un aumento comparable en la resistencia a la ciprofloxacina para mutantes derivados de NAM/ciprofloxacina, y la mayoría de los mutantes derivados de E. coli BW25113 mostraron un aumento de 2 a 4 veces en las MIC (Fig. 3A ). Sin embargo, dos mutantes, uno expuesto a atorvastatina/ciprofloxacino (BW_Ator_M1) y otro expuesto a ibuprofeno/ciprofloxacino (BW_Ibf_M1) mostraron niveles más altos de resistencia a ciprofloxacino con una CMI de 0,125 µg/mL (aumento de 8 veces). Otros dos mutantes derivados de E. coli BW25113 , acetaminofén/ciprofloxacino BW_Apap_M1 y BW_Apap_M2 mostraron un aumento de 16 y 4 veces en la resistencia a ciprofloxacino respectivamente. Curiosamente, los mutantes ibuprofeno/ciprofloxacino y acetaminofén/ciprofloxacino no solo mostraron un alto aumento en la CMI de ciprofloxacino, sino que las frecuencias de mutación para estas combinaciones NAM/ciprofloxacino fueron las únicas que fueron significativamente más altas que la frecuencia de mutación de ciprofloxacino solo. Por lo tanto, se seleccionaron estos mutantes para una evaluación adicional y se determinaron sus perfiles de resistencia contra un número amplio de antibióticos.
A , B Se seleccionó un número representativo (√n + 1) de mutantes derivados de E. coli de placas NAM/ciprofloxacina seleccionadas y se determinó la MIC de ciprofloxacina para E. coli BW25112 y E. coli 6146. APAP acetaminofeno, ATOR atorvastatina cálcica, CIP ciprofloxacina, DIC diclofenaco, FRS furosemida, IBF ibuprofeno, MET metformina, PSE pseudoefedrina, TMZ temazepam, TRA tramadol. * Indica mutantes seleccionados para un análisis posterior.
La evaluación de los mutantes derivados de E. coli 6146 y un examen de las CMI de ciprofloxacino revelaron una tendencia similar: los mutantes expuestos solo a ciprofloxacino mostraron un aumento de 4 a 8 veces en la CMI de ciprofloxacino en comparación con el tipo silvestre. Los mutantes expuestos a pseudoefedrina, temazepam y tramadol/ciprofloxacino no se investigaron más a fondo debido al número limitado de colonias en las placas. Cabe destacar que se observaron valores más altos de CMI de ciprofloxacino para los mutantes derivados de E. coli 6146 en comparación con los mutantes derivados de BW25113. La CMI de ciprofloxacino para un mutante expuesto a furosemida/ciprofloxacino (Frs_M1) aumentó a 0,5 µg/mL (aumento de 32 veces en la CMI), por lo que este mutante se seleccionó para una evaluación adicional. También se seleccionaron para un análisis posterior los mutantes Ator_M4 (expuesto a atorvastatina/ciprofloxacino) y Dic_M4 (expuesto a diclofenaco/ciprofloxacino) ya que estos también mostraron niveles más altos de resistencia a ciprofloxacino (CMI de 0,25 µg/mL) mostrando un aumento de 16 veces en comparación con WT E. coli 6146, así como un mutante acetaminofén/ciprofloxacino (6146_Apap_M2) con una CMI de 0,125 µg/mL, mostrando un aumento de 8 veces en la resistencia a ciprofloxacino. Finalmente, como se observó una mayor frecuencia de mutación para células después de la exposición a ibuprofeno/ciprofloxacino, un mutante representativo, 6146_Ibf_M1, que mostró una CMI de 0,06 µg/mL (aumento de 4 veces en la CMI) también se evaluó más a fondo.
La exposición a NAM aumenta la resistencia a múltiples antibióticos
La evaluación de mutantes seleccionados resistentes a ciprofloxacino de E. coli BW25113 y derivados de 6146 contra antibióticos selectos (Tabla S1 ), reveló una resistencia elevada a levofloxacino para un gran número de mutantes. Esto es esperado ya que los mismos mecanismos de resistencia son utilizados por E. coli contra estas quinolonas35 . Sin embargo, el aumento en los valores de MIC para ciprofloxacino o levofloxacino difirió para los mutantes evaluados aquí, lo que sugiere una ligera variación en los mecanismos de resistencia contra estos dos compuestos36 . De los compuestos evaluados aquí, además del aumento de las MIC de ciprofloxacino y levofloxacino, todos los mutantes derivados de BW25113 excepto BW_M1 y BW_Ator_M1 mostraron un aumento de 4 veces en la MIC para minociclina. También se observó un aumento de 4 veces en la MIC para ceftazidima para BW_Apap_M2 y BW_Ator_M1.
La evaluación de la resistencia de los mutantes del aislado clínico E. coli 6146 reveló valores de CMI más altos para ciprofloxacino en comparación con los mutantes derivados de la cepa E. coli BW25113. Por ejemplo, el mutante Frs_M1 vio un aumento de 32 veces para la CMI de ciprofloxacino y 16 veces para la CMI de levofloxacino. También se observó un aumento de 16 veces en la CMI de ciprofloxacino para los mutantes Dic_M4 y Ator_M4. Estos mutantes también mostraron una resistencia elevada a levofloxacino con un aumento de 8 veces en sus CMI de levofloxacino. Además, se observó una resistencia elevada contra cefepima en el mutante M1, Apap_M1, Dic_M1 y Ator_M1, con un aumento de 4 veces en la CMI de cefepima. Comparativamente, si bien los mutantes derivados de E. coli 6146 mostraron niveles más altos de resistencia a ciprofloxacino, levofloxacino y cefepima que la cepa BW25113, no se observó resistencia a otros antibióticos como ceftazidima y minociclina. Si bien la furosemida no incrementó la frecuencia de mutación, el mutante 6146_Frs_M1 mostró un aumento de 32 veces en la CMI de ciprofloxacino; por lo tanto, este mutante fue seleccionado para secuenciación.
La combinación de dos NAM aumenta la aptitud en presencia de una concentración sub-MIC de ciprofloxacino
Para explorar más a fondo los efectos de una combinación de medicamentos, se evaluaron las frecuencias de mutación en presencia de dos NAM y ciprofloxacino. Dado que la exposición de E. coli BW25113 y 6146 a concentraciones intestinales de ibuprofeno y acetaminofén, individualmente, aumentó significativamente las frecuencias de mutación, y dado que los mutantes derivados de la exposición a estos compuestos en presencia de ciprofloxacino mostraron valores elevados de CMI para ciprofloxacino y otras clases de antibióticos, se seleccionaron estos dos NAM para un análisis más detallado y se evaluó su capacidad para inducir mutaciones en presencia de un NAM adicional.
El crecimiento bacteriano se evaluó en presencia de combinaciones de NAM basadas en ibuprofeno y acetaminofén (Fig. 4 ). El crecimiento de E. coli BW25113 no se vio afectado significativamente, mientras que hubo un efecto modesto en el crecimiento de células de E. coli 6146 afectadas cuando se expusieron a dos NAM en ausencia de ciprofloxacino. Para E. coli BW25113, las combinaciones basadas en acetaminofén, con las excepciones de APAP + TMZ y APAP + MET, aumentaron la tasa de crecimiento, mientras que la mayoría de las combinaciones basadas en ibuprofeno disminuyeron la tasa de crecimiento. Para E. coli 6146, la adición de combinaciones de NAM, especialmente para las combinaciones de NAM basadas en ibuprofeno (excepto IBF + FRS), resultó en un aumento en la tasa de crecimiento, la densidad celular máxima y una reducción en el retraso de la fase logarítmica en comparación con el control sin NAM (3/4 CIP + DMSO). Los resultados indican que estas combinaciones de NAM podrían mejorar la aptitud y la adaptabilidad de estas dos cepas.
A – D El efecto de 2 NAMs en el crecimiento de E. coli BW25113 y E. coli 6146. E – H El efecto de 2 NAMS en el crecimiento de E. coli BW25113 y E. coli 6146 en presencia de ¾ MIC ciprofloxacino. El control de crecimiento representó células en ausencia de NAMs y ciprofloxacino. APAP acetaminofén, ATOR atorvastatina cálcica, CIP ciprofloxacino, DIC diclofenaco, FRS furosemida, IBF ibuprofeno, MET metformina, PSE pseudoefedrina, TMZ temazepam, TRA tramadol. Las curvas de crecimiento se realizaron cuatro veces en diferentes días con diferentes lotes de células. Aquí se muestran trazas representativas basadas en los resultados generales.
La combinación de dos NAM aumenta la frecuencia de mutación en presencia de ciprofloxacino
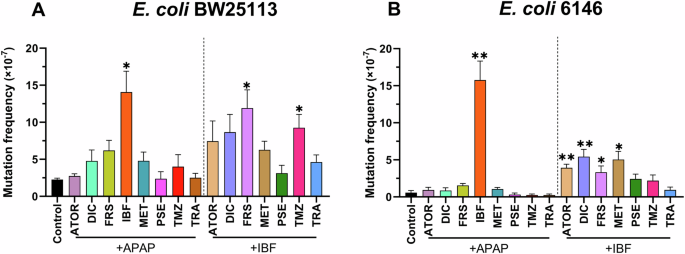
La evaluación de la frecuencia de mutación después de la exposición de células a 2 NAM y ciprofloxacino, reveló que la presencia de acetaminofeno combinado con ibuprofeno promovió significativamente el número de mutantes E. coli BW25113 ( P < 0,05) y 6146 ( P < 0,01) en comparación con las células expuestas solo a ciprofloxacino (Fig. 5 ). La frecuencia de mutación para E. coli expuesta a acetaminofén e ibuprofeno (1,40 ± 0,56 × 10 − 6 para BW25113 y 1,57 ± 0,51 × 10 − 6 para 6146) fue similar e incluso menor que la de las células expuestas a ibuprofeno solo (1,45 ± 0,19 × 10 − 6 para BW25113 y 2,30 ± 0,92 × 10 − 6 para 6146). Cabe destacar que también se observó una alta frecuencia de mutación ( P < 0,05) para las células BW25113 después de la exposición a ibuprofeno y temazepam. En cuanto a E. coli 6146, la exposición a atorvastatina, diclofenaco ( P < 0,01), furosemida y metformina ( P < 0,05) resultó en un aumento significativo de la frecuencia de mutación en comparación con las células expuestas solo al tratamiento con ciprofloxacino. Por el contrario, las células NAM restantes tratadas con ibuprofeno mostraron una menor frecuencia de mutación en comparación con las células expuestas a ibuprofeno.
A, B. Se indica la frecuencia de mutación tras la exposición a dos NAM para E. coli BW25113 y E. coli 6146. Las diferencias significativas entre los mutantes tratados con NAM adicionales y el grupo control (solo tratado con ciprofloxacino) se analizaron mediante pruebas t para muestras independientes y se corrigieron mediante el método de corrección de Bonferroni: * P < 0,05, ** P < 0,01 y **** P < 0,0001. APAP: acetaminofén; ATOR: atorvastatina cálcica; CIP: ciprofloxacino; DIC: diclofenaco; FRS: furosemida; IBF: ibuprofeno; MET: metformina; PSE: pseudoefedrina; TMZ: temazepam; TRA: tramadol.
Las combinaciones de NAM promueven la resistencia inducida por ciprofloxacina para producir fenotipos resistentes a la ciprofloxacina
Un número representativo (√n + 1) de mutantes derivados de E. coli BW25113 y 6146 se seleccionaron aleatoriamente de placas de combinación de ciprofloxacino/NAMs y se determinaron los valores de MIC para un panel de antibióticos (Fig. 6 ). Como se esperaba, la mayoría de los mutantes expuestos a dos NAMs/ciprofloxacino mostraron niveles más altos de resistencia a ciprofloxacino, con un aumento de 2 a 4 veces en los MIC en comparación con los mutantes expuestos a ciprofloxacino y un NAM. En mutantes derivados de E. coli BW25113, dos mutantes, BW_Apap/Ibf_M1 y BW_Ibf/Met_M1, mostraron un aumento de 32 veces en la resistencia a ciprofloxacino, con un MIC de 0,5 µg/mL. En particular, el mutante BW_Ibf/Dic exhibió los niveles más altos de resistencia a la ciprofloxacina, con una CMI de 1 µg/mL, lo que representa un aumento de 64 veces en comparación con el valor de CMI del WT.
A, B. Se seleccionó un número representativo (√n + 1) de mutantes derivados de E. coli de dos placas seleccionadas de NAM/ciprofloxacino y se determinó la CMI de ciprofloxacino para mutantes derivados de E. coli BW25112 y E. coli 6146. Los asteriscos indican los mutantes seleccionados para análisis posterior. APAP: acetaminofén; ATOR: atorvastatina cálcica; CIP: ciprofloxacino; DIC: diclofenaco; FRS: furosemida; IBF: ibuprofeno; MET: metformina; PSE: pseudoefedrina; TMZ: temazepam; TRA: tramadol.
Respecto a los mutantes derivados de E. coli 6146, dos mutantes, 6146_Ibf/Frs_M1 y 6146_Ibf/Dic_M1, mostraron una mayor resistencia a la ciprofloxacina, con CMI de 0,5 µg/mL y 0,25 µg/mL, mostrando un aumento de 32 y 16 veces en los valores de CMI de ciprofloxacina, respectivamente. Se seleccionaron los siguientes mutantes para evaluación adicional: 6146_Apap/Ibf_M1, 6146_Apap/Frs_M1, 6146_Apap/Ator_M1 y 6146_Ibf/Met_M1, de mutantes derivados de E. coli 6146, ya que se observaron CMI de 0,125 µg/mL.
Se seleccionaron mutantes con una resistencia a la ciprofloxacina al menos 8 veces mayor, representando cada grupo de aislados resistentes, y se determinaron sus perfiles de resistencia contra diez antibióticos (Tabla S2 ). Los mutantes derivados de la exposición a dos NAM exhibieron un mayor nivel de resistencia a la ciprofloxacina que los derivados de la exposición a un solo NAM, con mutantes como BW_Ibf/Dic_M1 cuya resistencia a la ciprofloxacina aumentó hasta 64 veces y los mutantes BW_Apap/Ibf_M1 y BW_Ibf/Met_M1 mostrando un aumento de 32 veces en la CMI de ciprofloxacina en relación con la E. coli BW25113 de tipo silvestre. De manera similar, los valores de CMI para levofloxacina, un antibiótico de la misma clase que la ciprofloxacina, también aumentaron de 4 a 16 veces para los mutantes derivados de NAM. En comparación, el aumento de la resistencia a los antibióticos de otras clases fue menor: los mutantes BW_Apap/Met_M1, BW_Ibf/Frs_M1 y BW_Ibf/Met_M2 mostraron un aumento de la CMI de 2 a 4 veces para minociclina, cefepima, ceftazidima y amoxicilina.
En el caso de E. coli 6146, los mutantes 6146_Ibf/Frs_M1 y 6146_Ibf/Dic_M1 presentaron valores de CMI de ciprofloxacino 32 y 16 veces superiores. Estos mutantes también mostraron una mayor resistencia a levofloxacino, con un aumento de la CMI de 16 y 8 veces, respectivamente. Finalmente, se observó una resistencia a cefepima 4 veces superior en los mutantes 6146_Ibf/Apap_M1 y 6146_Apap/Ator_M1, y este último también mostró una mayor resistencia a minociclina (con un aumento de la CMI de 4 veces).
El análisis genómico revela múltiples mutaciones en mutantes expuestos a NAM y ciprofloxacino
Para identificar las mutaciones que dieron lugar a la resistencia a la ciprofloxacina, se seleccionaron mutantes para la secuenciación del genoma completo. Los determinantes de resistencia evaluados se centraron principalmente en los implicados en la resistencia a las fluoroquinolonas, como GyrA/B, ParC/E 37 , 38 , así como en las bombas de eflujo RND (AcrAB-TolC) 39 , 40 , y sus reguladores (regulador local AcrR y regulador global MarR) 41 , ya que estos también desempeñan un papel importante en dicha resistencia 42 . Finalmente, también se evaluaron las mutaciones dentro de las proteínas de respuesta al estrés como SoxS y SoxR, ya que previamente se ha identificado que desempeñan un papel en la sobreexpresión de las bombas de eflujo 43 .
Varias mutaciones fueron identificadas en los mutantes evaluados, con mutaciones y deleciones observadas en MarR, AcrR y GyrA ( Tabla 2 ) . Para el mutante M1 derivado de BW25113, se detectó una mutación de sustitución única en SoxR (R20L). Mutaciones de GyrA descritas previamente, S83L, D87G y D87Y, que están asociadas supuestamente con una fuerte influencia en la resistencia a quinolonas36 , 44 fueron observadas en cinco mutantes. De estos, un mutante fue derivado de células de E. coli 6146 expuestas a furosemida y cuatro de células expuestas a ibuprofeno además de otro NAM y ciprofloxacino. Todas las mutaciones asociadas a la bomba de eflujo fueron localizadas principalmente en sus reguladores, AcrR y MarR. Entre estos, se identificó una mutación por sustitución de AcrR (T5N) en tres mutantes: Ibf_Frs y Apap_Met_M1 de BW25113, e Ibf/Apap_M1 de 6146. Asimismo, se observaron deleciones únicas en el regulador MarR entre estos mutantes. Curiosamente, muchos mutantes expuestos a dos NAM presentaron más de una mutación, es decir, se observó un aumento de la tasa de mutación (una estimación de la probabilidad de que ocurra una mutación por célula por generación), así como de la frecuencia de mutación.
Evaluación del papel de las bombas de eflujo en la resistencia adquirida
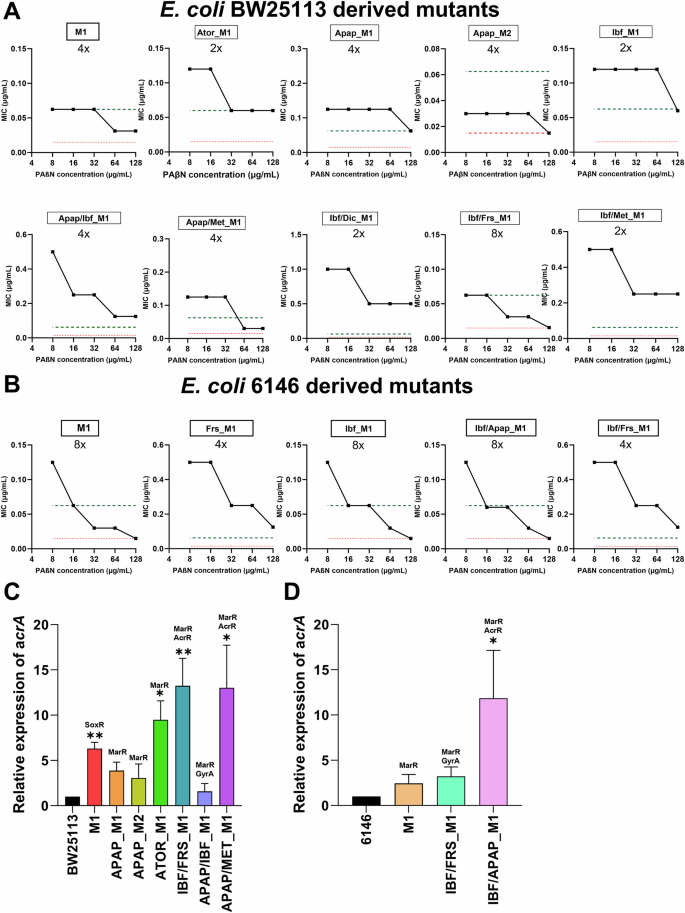
Dado que muchas mutaciones encontradas en los mutantes derivados de E. coli estaban relacionadas con reguladores de bombas de eflujo, se investigó el papel potencial de las bombas de eflujo RND en la resistencia observada a la ciprofloxacina. Se llevaron a cabo ensayos de tablero de ajedrez en presencia de PAβN, un inhibidor de la bomba de eflujo 45 , 46 , 47 . En total, se evaluaron 15 mutantes para determinar el papel de las bombas de eflujo RND en la resistencia, con resultados que revelaron que la adición de PAβN revirtió parcialmente la resistencia (reducción de 2 veces) en cuatro de los mutantes derivados de E. coli BW25113, con otros 5 mutantes que vieron una reducción de 4 veces en la CMI de ciprofloxacina y un mutante, BW_Ibf/Frs_M1, que mostró una reducción de 8 veces en la resistencia a la ciprofloxacina (Fig. 7A ). Se encontró que todos los mutantes derivados de BW25113 que mostraron una reducción de 4 a 8 veces en la resistencia a la ciprofloxacina presentaban mutaciones en MarR, AcrR o en ambos. Finalmente, la adición de PAβN a dos mutantes derivados de BW25113, BW_Apap_M2 y BW_Ibf/Frs_M1, eliminó la resistencia a la ciprofloxacina, lo que indica que la resistencia observada está mediada por el eflujo a través de las bombas de eflujo de tipo RND.
A, B Reversión de la resistencia en presencia del inhibidor de la bomba de eflujo PAβN para aislados mutantes derivados de E. coli BW25113 y E. coli 6146. Las líneas negras corresponden a las CMI en presencia de PAβN, y las líneas discontinuas rojas y verdes representan la CMI del tipo silvestre y la CMI promedio de mutantes expuestos solo a ciprofloxacino, respectivamente. C, D Los niveles de expresión de la bomba de eflujo AcrAB-TolC se evaluaron mediante RT-qPCR del gen acrA en mutantes derivados de E. coli BW25113 y E. coli 6146. Los resultados presentados son la media ± SEM de tres experimentos con diferentes lotes de células y preparaciones de ARN. Las diferencias significativas entre los mutantes y la cepa WT se analizaron mediante pruebas t de muestras independientes y se corrigieron mediante el método de corrección de Bonferroni: * P < 0,05, ** P < 0,01, *** P < 0,001 y **** P < 0,0001. APAP: acetaminofén; ATOR: atorvastatina cálcica; CIP: ciprofloxacino; DIC: diclofenaco; FRS: furosemida; IBF: ibuprofeno; MET: metformina.
La adición de PAβN a mutantes derivados de E. coli 6146 resultó en mayores reducciones en la resistencia, con mutantes 6146_Frs_M1 y 6146_Ibf/Frs_M1 mostrando reducciones de 4 veces en la MIC de ciprofloxacino, y una reducción de 8 veces en la resistencia observada en los tres mutantes restantes (Fig. 7B ). La adición de PAβN a tres mutantes derivados de E. coli 6146 revirtió la resistencia a ciprofloxacino a niveles de tipo salvaje. De estos, se encontró que 6146_Ibf_M1 albergaba una mutación GyrA (ΔS83), y ninguna mutación adicional asociada a la bomba de eflujo. Como tal, la reversión de la resistencia puede deberse a la regulación positiva transitoria de las bombas de eflujo 37 , 48 , que posteriormente fue inhibida por la adición de PAβN.
La RT-qPCR reveló la sobreexpresión de la bomba de eflujo AcrAB-TolC
Como se observaron numerosas mutaciones de MarR y AcrR en muchos de los mutantes de E. coli derivados de NAM y se observó una reducción en los valores de MIC en presencia del inhibidor de la bomba de eflujo PAβN, se realizó RT-qPCR para evaluar la regulación positiva de la bomba de eflujo AcrAB-TolC. Se seleccionaron para el análisis un total de diez mutantes de E. coli derivados de NAM, siete E. coli BW25113 y tres E. coli 6146 (Fig. 7 ). Se observó un aumento significativo en la expresión de la bomba de eflujo AcrAB en todos los mutantes que mostraron un aumento de veces que osciló entre 2,44 y 13,02 en comparación con sus cepas de tipo silvestre, con la excepción del mutante Apap/Ibf_M1 derivado de BW25113. El mutante Apap/Ibf_M1, derivado de BW25113, mostró una reducción de 4 veces en la CMI de ciprofloxacino tras la adición de PAβN y presentó una mutación en MarR (V84E), pero los niveles de expresión de AcrA solo aumentaron 1,56 veces. Cabe destacar que el aumento más sustancial en la expresión de AcrAB se observó en los mutantes Ibf/Frs_M1 ( P < 0,001), Apap/Met_M1 ( P < 0,001) e Ibf/Apap_M1 ( P < 0,01), derivados de BW25113, respectivamente, con una sobreexpresión de 13,24, 13,02 y 11,85 veces. Curiosamente, estos mutantes también presentaron mutaciones en MarR y AcrR (T5N). Además, el gen acrA que codifica la bomba de eflujo se reguló positivamente de forma significativa en el mutante Aror_M1 que contiene una eliminación de 2 pb en MarR ( P < 0,05).
Discusión
Se ha establecido claramente que los antibióticos en concentraciones subinhibitorias podrían promover el desarrollo y la propagación de la resistencia a los antibióticos 49 , 50 . De manera similar, se ha demostrado que los AINE, incluidos el acetaminofén y el ibuprofeno, inducen resistencia a los antibióticos en concentraciones altas (5 – 10 mM) al aumentar la expresión de la bomba de eflujo AcrAB-TolC 21 . La exposición frecuente a antidepresivos como la fluoxetina, la sertralina y la duloxetina podría inducir RAM a través de la adquisición de mutaciones 51 . Sin embargo, se sabe poco sobre la contribución de otros NAM a la aparición y propagación de la RAM y no se ha investigado el efecto de una variedad de NAM en concentraciones intestinales clínicamente relevantes. En general, el consumo a largo plazo de medicamentos entre las personas mayores conduce a una exposición combinada a antibióticos como la ciprofloxacina y los NAM en el rango de mg/L en la microbiota intestinal 7 , 52 . Nuestros hallazgos resaltan el potencial de E. coli para desarrollar mutaciones que conducen a RAM luego de la exposición a NAM y ciprofloxacino en RACF donde los medicamentos se usan ampliamente.
En general, nuestros resultados demostraron que dos analgésicos de consumo común (ibuprofeno y acetaminofén) pueden mejorar significativamente la resistencia a los antibióticos mediante mutación en concentraciones relevantes para el intestino en las cepas de E. coli BW25113 y E. coli 6146. Esto fue evidente en el mayor número de mutantes y la mejora de la densidad celular máxima. Otros NAM, como el diclofenaco y la furosemida, también aumentaron la frecuencia de mutación en comparación con el control sin NAM (ciprofloxacino con DMSO). Sin embargo, otros NAM como el temazepam, el tramadol y la pseudoefedrina tuvieron un impacto mínimo en la frecuencia de mutación para E. coli BW25113 y E. coli 6146. Aunque se sabe que la biodisponibilidad del ciprofloxacino se reduce significativamente cuando se coadministra con compuestos que contienen cationes metálicos o sucralfato 53 , no se observaron tales interacciones con los NAM utilizados en este estudio. Por el contrario, el diclofenaco aumentó la biodisponibilidad de la ciprofloxacina 54 , descartando la posibilidad de que su efecto en la frecuencia de mutación se debiera a la disminución de la biodisponibilidad de la ciprofloxacina. Considerando que la polifarmacia es común entre los ancianos en RACF, los efectos causados por el ibuprofeno y el acetaminofén también merecen atención. Curiosamente, encontramos que la exposición combinada a ibuprofeno y acetaminofén indujo una frecuencia de mutación similar a la del ibuprofeno solo, en lugar de resultar en un aumento acumulativo en la frecuencia de mutación. Sin embargo, los mutantes resultantes de la exposición a dos NAM adquiridos exhibieron niveles más altos de resistencia a la ciprofloxacina.
Además, los mutantes derivados de E. coli BW25113 y 6146 exhibieron una susceptibilidad reducida a múltiples antibióticos (≥4 veces), mostrando particularmente una resistencia aumentada a ciprofloxacino, con valores de MIC que aumentan a 32 veces para este agente. Entre estos antibióticos, los antibióticos β-lactámicos (amoxicilina, ceftazidima, meropenem), así como ciprofloxacino, levofloxacino y minociclina son sustratos de la bomba de eflujo AcrAB-TolC, que puede extrudir múltiples clases de antibióticos 55 , 56 . Por lo tanto, el aumento de la resistencia observada contra estos antibióticos sugiere un vínculo potencial con esta bomba de eflujo. Además, el análisis RT-qPCR reveló la sobreexpresión de la bomba de eflujo AcrAB-TolC, lo que respalda una relación estrecha entre los NAM y la expresión de la bomba de eflujo. En las primeras etapas del desarrollo de la resistencia a los antimicrobianos, las bombas de eflujo de fármacos desempeñan un papel fundamental al permitir que las bacterias sobrevivan a concentraciones iniciales subinhibitorias de antibióticos. Cuando una población bacteriana sensible se expone a niveles bajos de antibióticos, las bombas de eflujo pueden reducir transitoriamente las concentraciones intracelulares de fármacos, lo que permite que las células persistan y se adapten 57 . Esta ventana de supervivencia es crítica: proporciona tiempo para que las bacterias activen las respuestas al estrés y las redes reguladoras que regulan positivamente la expresión de la bomba de eflujo u otros mecanismos de resistencia. Estas bombas actúan de forma rápida e inespecífica, lo que las convierte en una de las primeras líneas de defensa contra los agentes antimicrobianos 58 . La sobreexpresión de las bombas de eflujo de fármacos conduce a la represión del gen de reparación de desajustes de ADN mutS y a un aumento concomitante de la frecuencia de mutación 59 , 60 . Por lo tanto, la sobreexpresión de la bomba de eflujo no solo favorece la supervivencia, sino que también facilita la selección de mutaciones genéticas estables que confieren una resistencia de alto nivel 61 . Por lo tanto, las bombas de eflujo no son meros transportadores pasivos, sino participantes activos en el panorama adaptativo de la evolución de la resistencia bacteriana. Por consiguiente, la capacidad de los NAM para inducir una mayor actividad de las bombas de eflujo, incluso sin causar mutaciones, podría tener un efecto considerable en la resistencia a los antimicrobianos (RAM). Además, en nuestro estudio, aunque la exposición a dos NAM no indujo mayores frecuencias de mutación, los mutantes derivados de dos combinaciones de NAM y ciprofloxacino mostraron mayores niveles de resistencia a múltiples antibióticos en comparación con los expuestos a una sola combinación de NAM y ciprofloxacino, debido al aumento de la tasa de mutación y a la inducción de múltiples mutaciones por colonia.
Se realizó la secuenciación completa del genoma para dilucidar los mecanismos responsables de la resistencia a múltiples antibióticos en mutantes. El análisis reveló que la combinación de NAM con ciprofloxacino puede inducir polimorfismos de un solo nucleótido (SNP) no sinónimos en genes reguladores, lo que lleva a la sobreexpresión de la bomba de eflujo. Además de las mutaciones que ocurren en las regiones determinantes de resistencia a quinolonas en genes de topoisomerasas como gyrA , también se identificaron múltiples mutaciones (deleciones y sustituciones) en genes reguladores, como marR , acrR y soxR que son reguladores del sistema AcrAB-TolC, destacando la conexión entre los antibióticos y las bombas de eflujo 50 , 62 . MarR regula negativamente el factor de transcripción MarA, que a su vez regula la expresión de muchas otras proteínas, incluidas las de la bomba de eflujo AcrAB-TolC. Por lo tanto, las mutaciones en MarR resultaron en una mayor expresión del operón marRAB, lo que conduce a la regulación positiva de la bomba de eflujo AcrAB-TolC y reduce la susceptibilidad a los antibióticos 63 . El mutante BW_APAP/IBF que lleva MarR V84E y GyrA Δ83 no solo confirió alta resistencia a la ciprofloxacina, sino que también tuvo un pequeño aumento en las MIC a ceftazidima y minociclina. Esto fue consistente con un estudio previo de que los aislados clínicos que llevan MarR V84E proporcionaron resistencia a los antibióticos a diferentes clases de antibióticos 64 . AcrR actúa como un represor local para prevenir la sobreexpresión de la bomba de eflujo AcrAB-TolC. Sorprendentemente, tras la exposición a dos NAM/ciprofloxacina, se observó un aumento en el número de mutaciones en ciertos aislados de E. coli , con cinco mutantes que desarrollaron mutaciones tanto en MarR como en AcrR o en GyrA y MarR. De estos, la mutación T5N observada en AcrR ocurrió en tres mutantes, que mostraron una susceptibilidad reducida a diferentes clases de antibióticos. Además, el análisis RT-qPCR mostró que los mutantes que portaban la sustitución T5N dieron como resultado el nivel más alto de expresión de la bomba de eflujo AcrAB-TolC. Un estudio previo postuló que la mutación T5N observada en AcrR puede hacer que este regulador sea inactivo o potencialmente desestabilizarlo 65 , con el aumento de la expresión de la bomba de eflujo AcrAB-TolC observado en este estudio, lo que respalda esta teoría. Algunos reguladores globales, como SoxS, están regulados por SoxR, cuya activación por superóxidos contribuye a la expresión de SoxS, que a su vez regula positivamente la transcripción de acrAB 66 , 67 . La sustitución R20L observada en el mutante BW_M1 podría explicar la regulación positiva de la bomba de eflujo AcrAB-TolC, lo que conduce a una mayor resistencia a los antibióticos.
Nuestros hallazgos basados en el análisis fenotípico revelaron que solo unos pocos mutantes revierten completamente la resistencia a la ciprofloxacina a los niveles originales en presencia de PAβN, un inhibidor de la bomba de eflujo. Se ha demostrado que los altos niveles de resistencia a los antibióticos no solo se atribuyen a una mayor expresión de las bombas de eflujo, sino que también pueden resultar de la acumulación de mutaciones en diferentes dianas 41 , 42 , 68. En aislados de E. coli resistentes a las fluoroquinolonas , las mutaciones iniciales en gyrA , como S83L, que es la sustitución más común, son esenciales para el desarrollo de una resistencia de alto nivel a las fluoroquinolonas 69. Un alto nivel de resistencia a las fluoroquinolonas es un proceso gradual y a menudo es el resultado de la acumulación de sustituciones de aminoácidos 70. En este estudio, una combinación de mutaciones de gyrA (S83L y D87G/Y) junto con mutaciones en los reguladores de la bomba de eflujo contribuyeron a los altos niveles de resistencia a las fluoroquinolonas. Los microbios aumentan el eflujo de antimicrobianos para reducir su concentración intracelular y proporcionarles aptitud biológica, lo que les permite sobrevivir más tiempo de lo esperado en presencia de bajas concentraciones de antibióticos 41 , 63. Además, las mutaciones en aislados clínicos tienden a favorecer a aquellos que imponen un pequeño coste a la aptitud biológica y confieren una pequeña reducción en la susceptibilidad a los antibióticos. Esta ventaja de crecimiento permite que la cepa acumule mutaciones gradualmente, evolucionando finalmente hacia una resistencia de alto nivel 41 , 63 .
Algunos NAM exhiben toxicidad contra E. coli , alterando la fisiología bacteriana, la respuesta al estrés y el crecimiento 71 . Sin embargo, en ausencia de antibióticos, la exposición a largo plazo a NAM por sí sola puede no ejercer suficiente presión selectiva para impulsar la resistencia cruzada a los antibióticos. Este fenómeno cambia cuando E. coli se expone tanto a antibióticos como a NAM, con estudios previos que informan un efecto sinérgico entre ellos. Por ejemplo, los antidepresivos como la sertralina en combinación con tetraciclina y la duloxetina con cloranfenicol inducen resistencia múltiple a los antibióticos en E. coli 52 , 72 . En nuestro estudio, la coexistencia de ciprofloxacino y NAM conduce a la evolución de la resistencia cruzada a los antibióticos. Dado que la exposición generalizada tanto a NAM como a ciprofloxacino es común entre los residentes en RACF, el desarrollo de RAM en E. coli in vivo parece plausible.
La polifarmacia no solo aumenta el riesgo de efectos adversos de los medicamentos y la probabilidad de interacciones medicamentosas, sino que también complica el uso de antibióticos. En particular, se ha demostrado que varios productos farmacéuticos de consumo común, como los inhibidores de la bomba de protones (IBP) y la metformina, causan disbiosis intestinal al alterar la composición de la microbiota intestinal. La erradicación de la mayoría de la microbiota intestinal protectora permite un entorno propicio para el crecimiento excesivo y la colonización de bacterias dañinas como Clostridium difficile 5 , 73 . Además, se ha observado que, más allá de la mayoría de los antibióticos conocidos, el uso de inhibidores de la bomba de protones (IBP) también aumenta el riesgo de infecciones por Clostridium difficile 74 . Como se esperaba, la incidencia de infecciones por Clostridium difficile se duplicó cuando los IBP se administraron simultáneamente con fluoroquinolonas 75 . De manera similar, nuestro estudio revela que ciertos NAM en concentraciones intestinales relevantes pueden promover la RAM en presencia de ciprofloxacino. Estos hallazgos resaltan la necesidad urgente de reevaluar las combinaciones entre NAM y antibióticos, particularmente dentro de regímenes de polifarmacia complejos.
Colectivamente, este estudio proporciona evidencia de que la medicación utilizada para aliviar el dolor, como el acetaminofén y el ibuprofeno, cuando se combina con ciprofloxacino, no solo aumenta significativamente la frecuencia de mutación sino que también confiere resistencia a múltiples antibióticos. Otros NAM como el diclofenaco y la furosemida también contribuyen al aumento de la frecuencia de mutación y a los altos niveles de resistencia a las fluoroquinolonas. Aunque la frecuencia de mutación inducida por dos NAM no es sinérgica para el aumento de la frecuencia de mutación, se observa una resistencia de alto nivel a la ciprofloxacino entre los mutantes debido a una mayor tasa de mutación y la posterior acumulación de mutaciones. Las personas mayores, como las que viven en RACF, toman numerosos medicamentos a diario 76, lo que destaca la necesidad de considerar las combinaciones de medicamentos al recetar. Este estudio también allana el camino para investigar el efecto de los regímenes que comprenden múltiples medicamentos (es decir, polifarmacia) en el desarrollo de RAM.
Métodos
Cepas bacterianas y condiciones de cultivo
Las cepas bacterianas utilizadas en este estudio incluyeron la cepa E. coli BW25113, un derivado de la cepa E. coli K-12 77 , 78 , y E. coli 6146, una cepa sensible a los antibióticos aislada de una muestra fecal obtenida de una comunidad de jubilados en Adelaida, Australia. El proyecto recibió la aprobación ética del comité de ética de investigación humana de la Universidad del Sur de Australia para recolectar muestras fecales de RACF y residentes jubilados (ID de la aplicación: 201882). Típicamente, las cepas se cultivaron en medios Luria-Bertani (LB) (Difco®, Franklin Lakes, Nueva Jersey) a 37 °C. Los NAM se disolvieron en dimetilsulfóxido (DMSO) y los antibióticos se disolvieron en sus solventes recomendados. Se eligió agar nutritivo (NA) como medio para los estudios, con placas de NA suplementadas con ciprofloxacino y NAM para evaluar la frecuencia de mutación.
Condiciones de cultivo y ensayos de crecimiento
Los ensayos de crecimiento se realizaron en un volumen de 200 μL del inóculo en caldo Mueller Hinton II ajustado catiónicamente (BD, Australia). Brevemente, se añadieron células estandarizadas a una DO₃₄ de 0,015 a cada pocillo de una placa de microtitulación de 96 pocillos (Costar®) que contenía rangos de concentración de ciprofloxacino (0, ½ y ¾ de la CMI de ciprofloxacino) y cada NAM a una concentración intestinal de 7. La placa de 96 pocillos se incubó durante 20 horas a 37 °C en el lector multietiqueta EnSpire 2300 (PerkinElmer, EE. UU.). Se midió y registró la absorbancia a 600 nm a intervalos de 20 minutos.
Estimación de la concentración de NAM en el intestino
La concentración intestinal de NAM se calculó mediante un modelo basado en Maier et al. 7. En resumen, este método supone que un fármaco oral se disuelve en 300 ml de agua, incluyendo el agua necesaria para tragar la pastilla (240 ml) y el agua restante (60 ml) en el intestino delgado. El modelo también supone que el 90 % de los fármacos orales se absorbe en el torrente sanguíneo, lo que significa que el 10 % permanece en el intestino. Las dosis individuales se obtendrán de la base de datos de Dosis Diarias Definidas (DDD) de la OMS 79 .
Con base en el modelo anterior, las concentraciones de NAM utilizadas en el estudio fueron 216 μg/mL para acetaminofeno, 15,4 μg/mL para diclofenaco, 13,2 μg/mL para furosemida, 99,8 μg/mL para ibuprofeno, 194,6 μg/mL para metformina, 6,6 μg/mL para atorvastatina, 40 μg/mL para pseudoefedrina, 3,3 μg/mL para temazepam y 33,3 μg/mL para tramadol.
Pruebas de susceptibilidad a los antimicrobianos
Los antimicrobianos evaluados en este estudio incluyeron ciprofloxacino, norfloxacino, levofloxacino, minociclina, cefepima, meropenem, eritromicina y novobiocina, todos obtenidos de Sigma Aldrich (Sídney, Australia). También se utilizaron paracetamol, furosemida, atorvastatina cálcica, diclofenaco, ibuprofeno y metformina, obtenidos de Sigma Aldrich (Sídney, Australia).
Las concentraciones inhibitorias mínimas (CIM) se determinaron mediante el ensayo de microdilución en caldo ISO 20776-1, según lo recomendado por el Comité Europeo de Pruebas de Susceptibilidad a los Antimicrobianos 80 . Cada cepa se analizó por triplicado.
Determinación de la frecuencia de mutación
La frecuencia de mutación se evaluó utilizando tanto E. coli BW25113 (tipo salvaje) como E. coli 6146 (un aislado sensible a antibióticos de la muestra fecal de un residente jubilado). Cultivos nocturnos cultivados a 37 °C en 5 mL de caldo LB se diluyeron (1:100) en LB y se cultivaron hasta la fase logarítmica media (DO 600 de 0,4-0,6) a 37 °C. Un volumen de 100 μL de cada cultivo se extendió sobre placas de NA que contenían 1x, 2x y 4x la MIC de ciprofloxacino (0,015 a 0,06 μg/mL) y NAM, a concentraciones clínicamente relevantes para seleccionar bacterias resistentes a ciprofloxacino. Las placas que contenían ciprofloxacino y el disolvente NAM (DMSO) sirvieron como control solo de ciprofloxacino. Los cultivos se incubaron durante 48 h a 37 °C 7 . La frecuencia de mutación (número de mutantes en la población) se calculó según la siguiente ecuación 81 :
Para corregir el sesgo de siembra, se realizaron ensayos en cuatro días diferentes con distintos lotes de células. Se seleccionó aleatoriamente un número representativo de mutantes derivados de E. coli , calculado como √n + 1 (donde n es el número de colonias por placa), para la determinación de la CMI de ciprofloxacino. Los mutantes representativos que mostraron una resistencia a ciprofloxacino al menos cuatro veces mayor se seleccionaron para análisis posteriores, evitando así la inclusión de cepas cuyo aumento de la CMI pudiera deberse a la variación en el ensayo de CMI.
Ensayos de tablero de ajedrez
Para determinar el efecto de la resistencia mediada por la bomba de eflujo de RND, se llevaron a cabo ensayos de tablero de ajedrez en aislados resistentes a ciprofloxacino en presencia y ausencia del inhibidor de la bomba de eflujo fenilalanina-arginina β-naftilamida (PAβN) como se describió previamente 82 , 83 , 84 . La ciprofloxacina se diluyó verticalmente hacia abajo en la placa de microtitulación de 96 pocillos (Costar®) (diluciones seriadas de 2 veces), y PAβN se diluyó seriadamente horizontalmente a través de la placa (diluciones seriadas de 2 veces). Se añadió un inóculo de 1,5 × 106 UFC/mL a cada pocillo y las placas se incubaron a 37 °C durante 18 horas, agitando a 120 rpm. Las MIC en presencia de PAβN se utilizaron para comparar el cambio en las MIC en ausencia de PaβN.
Extracción de ADN, secuenciación del genoma completo y procesamiento de datos
El ADN genómico se extrajo con el kit de extracción de ADN microbiano NucleoSpin® (MACHEREY-NAGEL, Alemania), siguiendo las instrucciones del fabricante. La concentración y la pureza del ADN (A₂₆ / A₂₆ , 1,8-2,0) se evaluaron con el lector de placas Cytation 5® (Bio-Tek®, Winooski, EE. UU.) y la calidad del ADN se analizó mediante electroforesis en gel de agarosa. Todas las muestras se enviaron al Centro Australiano de Investigación Genómica (AGRF) para la preparación de la biblioteca y la secuenciación de extremos emparejados (paired-end) se realizó en la plataforma Illumina Hi-Seq.
Las lecturas de secuenciación de pares terminales de 150 bases se ensamblaron y anotaron mediante el proceso de desarrollo TORMES (v.1.3) 85 . Para evaluar posibles mutaciones, se compararon los borradores del genoma de mutantes derivados de E. coli con el de tipo silvestre mediante BRESEQ 86 . Los aminoácidos de las proteínas diana de tipo silvestre se alinearon con las secuencias de aminoácidos de los mutantes mediante CLUSTAL O v.1.2.4 87 . Las variaciones de secuencia se visualizaron mediante ESPript v.3.0 88 .
Extracción de ARN, reacción en cadena de la polimerasa cuantitativa en tiempo real con transcripción inversa (RT-qPCR)
Se realizó una reacción en cadena de la polimerasa cuantitativa en tiempo real (RT-qPCR) con transcripción inversa para cuantificar el nivel transcripcional de acrA , que codifica la proteína de fusión de membrana AcrA de la bomba de eflujo multifármaco AcrAB-TolC. Se seleccionaron aislados con una resistencia a la ciprofloxacina más de cuatro veces superior (CMI de ciprofloxacina ≥ 0,015 μg/mL) para el análisis mediante RT-qPCR con el fin de evaluar el posible papel de las bombas de eflujo en esta resistencia.
Los cultivos nocturnos inoculados con colonias individuales se diluyeron (1:100) en caldo LB y se cultivaron hasta la fase logarítmica media (A₂₄ de 0,5) a 37 °C. El ARN total se extrajo como se describió previamente 42 . Brevemente, se centrifugó 1 ml de cultivos, se resuspendió en Trizol y cloroformo, y luego se purificó aún más utilizando el kit de extracción de ARN NucleoSpin® (MACHEREY-NAGEL, Alemania), seguido de una digestión con rDNasa, que se realizó para eliminar el ADN durante este período. La concentración y la pureza del ARN total se cuantificaron mediante Cytation 5® (Bio-Tek®, Winooski, EE. UU.).
Todas las reacciones de RT-qPCR se realizaron utilizando un ciclador de inducción magnética (Bio Molecular Systems, Australia) y el kit KAPA SYBR® FAST qPCR Master Mix (Kapa Biosystems, EE. UU.). El gen constitutivo, recA , se utilizó para normalizar los niveles transcripcionales de acrA , el gen diana. Las secuencias del cebador incluyen AcrA_F 5′-GATCCAGCCACCTATCAGGC-3′; AcrA_R 5′-ACGCTTGACCGTCAGTTGAT-3′; RecA_F 5′- TTAAACAGGCCGAATTCCAG-3′; RecA_R 5′-CCGCTTTCTCAATCAGCTTC-3′. Los parámetros de ciclado fueron los siguientes: transcripción inversa a 42 °C durante 10 min, inactivación a 95 °C durante 3 min y 40 ciclos de PCR a 95 °C durante 5 s, 60 °C durante 30 s y 72 °C durante 15 s. No se estableció control de plantilla (NTC) ni control de transcriptasa inversa (NRT), y las muestras de reacción de qPCR se realizaron por triplicado.
Análisis estadístico
Se utilizó Graph Prism v9 para todos los análisis de datos. Las diferencias significativas se evaluaron mediante pruebas t para muestras independientes y se corrigieron mediante el método de Bonferroni: * p < 0,05, ** p < 0,01, *** p < 0,001 y **** p < 0,0001. Todos los experimentos se realizaron por triplicado.
Disponibilidad de datos
La secuenciación se ha depositado en la base de datos NCBI BioProject PRJNA1281333.
Referencias
-
Plan de acción mundial de la OMS sobre la resistencia a los antimicrobianos. Microbe Mag. 10 , 354–355 (2015).
-
Murray, JL et al. Carga mundial de resistencia bacteriana a los antimicrobianos en 2019: un análisis sistemático. Lancet 399 , 629–655 (2022).
-
Rousham, EK, Unicomb, L. e Islam, MA. Factores humanos, animales y ambientales que contribuyen a la resistencia a los antibióticos en entornos de bajos recursos: integración de enfoques conductuales, epidemiológicos y de Una Sola Salud. Proc. R. Soc. B: Biol. Sci. 285 , 20180332 (2018).
-
Martin, MJ, Thottathil, SE, y Newman, TB: Uso excesivo de antibióticos en la ganadería: un llamado a la acción para los profesionales de la salud. En (Vol. 105, págs. 2409-2410): Asociación Americana de Salud Pública. (2015)
-
Imhann, F. et al. Los inhibidores de la bomba de protones afectan el microbioma intestinal. Gut 65 , 740–748 (2016).
-
Wang et al. Los fármacos no antibióticos mejoran la transmisión de genes exógenos de resistencia a antibióticos mediante la transformación bacteriana. ISME J. 14 , 2179–2196 (2020).
-
Maier, L. et al. Impacto significativo de fármacos no antibióticos en las bacterias intestinales humanas. Nature 555 , 623–628 (2018).
-
Hamad, B. El mercado de antibióticos. Nat. Rev. Drug Discov. 9 , 675–676 (2010).
-
Mikulic, M. Industria farmacéutica global: estadísticas y datos . Recuperado de https://www.statista.com/topics/1764/global-pharmaceutical-industry/#topicOverview . (2024).
-
Christensen, K., Doblhammer, G., Rau, R. y Vaupel, J. W. Envejecimiento de las poblaciones: los desafíos futuros. Lancet 374 , 1196–1208 (2009).
-
Sluggett, JK, Inacio, MC y Caughey, GE. Gestión de medicamentos en cuidados a largo plazo: uso de evidencia generada a partir de datos del mundo real para impulsar cambios en las políticas en el ámbito australiano. Am. J. Epidemiol. 193 , 1645–1649 (2024).
-
Wastesson, JW, Morin, L., Tan, EC y Johnell, K. Actualización sobre las consecuencias clínicas de la polifarmacia en adultos mayores: una revisión narrativa. Opinión de expertos. Drug Saf. 17 , 1185–1196 (2018).
-
Dagli, RJ y Sharma, A. Polifarmacia: un factor de riesgo global para las personas mayores. J. Int Oral. Health 6 , i–ii (2014).
-
Wang, KN et al. ¿Cuál es la mejor definición de polifarmacia para predecir caídas, hospitalizaciones y mortalidad en centros de atención a largo plazo? J. Am. Med. Dir. Assoc. 22 , 470–471 (2021).
-
Sluggett, JK et al. Tendencias nacionales en el uso de antibióticos en residencias geriátricas australianas, 2005-2016. Clin. Infect. Dis. 72 , 2167-2174 (2021).
-
Nicolle, LE et al. Uso de antimicrobianos en centros de cuidados a largo plazo. Infect. Control Hosp. Epidemiol. 21 , 537–545 (2000).
-
Fleming, A., Bradley, C., Cullinan, S. y Byrne, S. Prescripción de antibióticos en centros de atención a largo plazo: una metasíntesis de investigación cualitativa. Drugs Aging 32 , 295–303 (2015).
-
Maher, D. et al. Vigilancia del uso de antimicrobianos en centros de atención a largo plazo: un estudio de mapeo de antimicrobianos. J. Am. Med. Dir. Assoc. 25 , 105144 (2024).
-
ACSQHC. Uso de antimicrobianos en la comunidad: 2023. Recuperado de la Comisión Australiana de Seguridad y Calidad en la Atención Sanitaria: https://www.safetyandquality.gov.au/publications-and-resources/resource-library/antimicrobial-use-community-2023 (2024).
-
Ko, HHT, Lareu, RR, Dix, BR y Hughes, JD Estatinas: ¿destructoras o generadoras de resistencia a los antimicrobianos?. PeerJ 5 , e3952 (2017).
-
Verma, T. et al. Los antiinflamatorios no esteroideos, acetaminofén e ibuprofeno, inducen resistencia fenotípica a antibióticos en Escherichia coli: funciones de marA y acrB. FEMS Microbiol. Lett. 365 , fny251 (2018).
-
Bírošová, L., Lépesová, K., Grabic, R. y Mackuľak, T. Los fármacos no antimicrobianos pueden afectar el desarrollo de resistencia a los antibióticos en aguas residuales hospitalarias. Environ. Sci. Pollut. Res. 27 , 13501–13511 (2020).
-
Roughead, EE et al. Efecto de un servicio farmacéutico continuo para reducir el deterioro y las reacciones adversas inducidos por medicamentos en residencias de ancianos: un ensayo controlado aleatorizado multicéntrico (ensayo ReMInDAR). Age ageing 51 , afac092 (2022).
-
Plan, Informe de Gastos y Prescripciones del PB PBS, del 1 de julio de 2023 al 30 de junio de 2024. Canberra: Departamento de Salud del Gobierno de Australia , 2020-2021. (2025)
-
Breidenstein, EB, Bains, M. y Hancock, RE. Participación de la proteasa Ion en la respuesta SOS desencadenada por ciprofloxacino en Pseudomonas aeruginosa PAO1. Agentes antimicrobianos Chemother. 56 , 2879–2887 (2012).
-
Blondeau, JM. Cuestiones actuales en el tratamiento de las infecciones del tracto urinario: ciprofloxacino de liberación prolongada como una nueva opción terapéutica. Drugs 64 , 611–628 (2004).
-
Fasugba, O., Gardner, A., Mitchell, BG y Mnatzaganian, G. Resistencia a la ciprofloxacina en infecciones urinarias por Escherichia coli adquiridas en la comunidad y en el hospital: una revisión sistemática y un metaanálisis de estudios observacionales. BMC Infect. Dis. 15 , 1–16 (2015).
-
Juurlink, DN et al. Interacciones farmacológicas en pacientes ancianos hospitalizados por toxicidad farmacológica. JAMA 289 , 1652–1658 (2003).
-
Kalisch Ellett, LM et al. Ingresos hospitalarios relacionados con la medicación en residentes de residencias de ancianos. Australas. J. Ageing 40 , e323–e331 (2021).
-
Rodríguez-Martínez, J. et al. Correlación de los niveles de resistencia a quinolonas y diferencias en la expresión basal e inducida por quinolonas de tres plásmidos que contienen qnrA. Clin. Microbiol. Infect. 12 , 440–445 (2006).
-
Mulder, M. et al. Factores de riesgo de resistencia a la ciprofloxacina en infecciones urinarias adquiridas en la comunidad por Escherichia coli en una población de edad avanzada. J. Antimicrob. Chemother. 72 , 281–289 (2016).
-
Sapula, SA et al. Alcance de la resistencia a los antimicrobianos en residencias de ancianos, determinado mediante el análisis de Escherichia coli y el resistoma total de aguas residuales. Microbiol. Spectr. 11 , e00731–00723 (2023).
-
Stuart, R. et al. Uso y abuso de antibióticos en residencias para personas mayores. Intern. Med. J. 42 , 1145–1149 (2012).
-
Visvalingam, J., Palaniappan, K. y Holley, RA. Mejora in vitro de la susceptibilidad a antibióticos de Escherichia coli resistente a fármacos mediante cinamaldehído. Food Control 79 , 288–291 (2017).
-
Morgan-Linnell, SK, Becnel Boyd, L., Steffen, D. y Zechiedrich, L. Mecanismos responsables de la resistencia a fluoroquinolonas en aislamientos clínicos de Escherichia coli. Antimicrobiano. Chemother. 53 , 235–241 (2009).
-
Fu, Y. et al. Patrones específicos de mutaciones en gyr A determinan la diferencia de resistencia a ciprofloxacino y levofloxacino en Klebsiella pneumoniae y Escherichia coli . BMC Infect. Dis. 13 , 8 (2013).
-
Arzanlou, M., Chai, WC y Venter, H. Resistencia antimicrobiana intrínseca, adaptativa y adquirida en bacterias gramnegativas. Essays Biochem. 61 , 49–59 (2017).
-
Redgrave, LS, Sutton, SB, Webber, MA y Piddock, LJ. Resistencia a las fluoroquinolonas: mecanismos, impacto en las bacterias y rol en el éxito evolutivo. Trends Microbiol. 22 , 438–445 (2014).
-
Lomovskaya, O. y Bostian, KA. Aplicaciones prácticas y viabilidad de los inhibidores de la bomba de eflujo en la clínica: una visión para su uso aplicado. Biochem. Pharmacol. 71 , 910–918 (2006).
-
Venter, H., Mowla, R., Ohene-Agyei, T. y Ma, S. Bombas de eflujo de fármacos de tipo RND de bacterias gramnegativas: mecanismo molecular e inhibición. Front. Microbiol. 6 , 377 (2015).
-
Piddock, L. J. Bombas de eflujo de resistencia a múltiples fármacos, codificadas cromosómicamente y clínicamente relevantes en bacterias. Clin. Microbiol. Rev. 19 , 382–402 (2006).
-
Amsalu, A. et al. Resistencia cruzada a antibióticos y biocidas impulsada por bombas de eflujo en Pseudomonas aeruginosa aisladas de diferentes nichos ecológicos: un estudio de caso sobre el desarrollo de resistencia a múltiples fármacos en zonas de alto riesgo ambiental. Microorganisms 8 , 1647 (2020).
-
Koutsolioutsou, A., Pena-Llopis, S. y Demple, B. Las mutaciones constitutivas de soxR contribuyen a la resistencia a múltiples antibióticos en aislamientos clínicos de Escherichia coli. Antimicrobiano. Chemother. 49 , 2746–2752 (2005).
-
Bagel, S., Hüllen, V., Wiedemann, B. y Heisig, P. Impacto de las mutaciones gyrA y parC en la resistencia a quinolonas, el tiempo de duplicación y el grado de superenrollamiento de Escherichia coli. Antimicrob. Agents Chemother. 43 , 868–875 (1999).
-
Lomovskaya, O. et al. Identificación y caracterización de inhibidores de bombas de eflujo de resistencia a múltiples fármacos en Pseudomonas aeruginosa : nuevos agentes para terapia combinada. Antimicrob. Agents Chemother. 45 , 105–116 (2001).
-
Venter, H. Revertir la resistencia para contrarrestar la resistencia a los antimicrobianos en la prioridad crítica de la Organización Mundial de la Salud sobre los patógenos más peligrosos. Biosci. Rep. 39 , BSR20180474 (2019).
-
Kinana, AD, Vargiu, AV, May, T. y Nikaido, H. Aminoacil β-naftilamidas como sustratos y moduladores de la bomba de eflujo multifármaco AcrB. Proc. Natl. Acad. Sci. 113 , 1405–1410 (2016).
-
Hernando-Amado, S. et al. Bombas de eflujo multifármaco como actores principales en la resistencia intrínseca y adquirida a los antimicrobianos. Drug Resist. Actualizaciones 28 , 13–27 (2016).
-
Gullberg, E. et al. Selección de bacterias resistentes a concentraciones muy bajas de antibióticos. PLoS Pathog. 7 , e1002158 (2011).
-
Sanz-García, F. et al. Concentraciones bajas de ciprofloxacino seleccionan mutantes multirresistentes que sobreproducen bombas de eflujo en aislados clínicos de Pseudomonas aeruginosa. Microbiol. Spectr. 10 , e00723–00722 (2022).
-
Wang, Y. et al. Los antidepresivos pueden inducir mutación y aumentar la persistencia frente a múltiples antibióticos. Proc. Natl. Acad. Sci. 120 , e2208344120 (2023).
-
Shi, D. et al. La exposición combinada a fármacos no antibióticos y antibióticos en el intestino promueve sinérgicamente el desarrollo de multirresistencia en Escherichia coli. Gut Microbes 14 , 2018901 (2022).
-
Issa, MM et al. Efectos del paracetamol en la farmacocinética de la ciprofloxacina plasmática mediante un ensayo microbiológico. Clin. Drug Investig. 27 , 463–467 (2007).
-
Iqbal, Z., Khan, A., Naz, A., Khan, JA y Khan, GS Interacción farmacocinética de ciprofloxacino con diclofenaco. (2009).
-
Kobylka, J. et al. AcrB: una máquina de eflujo de fármacos eficaz y eficiente. Ann. NY Acad. Sci. 1459 , 38–68 (2020).
-
Du, D. et al. Estructura de la bomba de eflujo multifármaco AcrAB-TolC. Nature 509 , 512–515 (2014).
-
Du, D., Venter, H., Pos, KM, y Luisi, BF. La maquinaria y el mecanismo del eflujo de múltiples fármacos en bacterias gramnegativas . Caister Academic Press, (2013).
-
Du, D. et al. Bombas de eflujo multifármaco: estructura, función y regulación. Nat. Rev. Microbiol. 16 , 523–539 (2018).
-
El Meouche, I. y Dunlop, MJ. La heterogeneidad en la expresión de la bomba de eflujo predispone a las células resistentes a los antibióticos a la mutación. Science 362 , 686–690 (2018).
-
Darby, EM et al. Mecanismos moleculares de la resistencia a los antibióticos revisados. Nat. Rev. Microbiol. 21 , 280–295 (2023).
-
Grimsey, EM et al. La sobreexpresión de RamA, que regula la producción de la bomba de eflujo de resistencia a múltiples fármacos AcrAB-TolC, aumenta la tasa de mutación e influye en el fenotipo de resistencia a fármacos. Antimicrobiano. Agentes Chemother. 64 , e02460–19 (2020).
-
Bialek-Davenet, S. et al. Selección in vitro de mutantes ramR y soxR que sobreexpresan sistemas de eflujo mediante fluoroquinolonas y cefoxitina en Klebsiella pneumoniae. Antimicrobiano Chemother. 55 , 2795–2802 (2011).
-
Praski Alzrigat, L., Huseby, DL, Brandis, G. y Hughes, D. El coste de adaptación restringe el espectro de mutaciones de marR en Escherichia coli resistente a la ciprofloxacina. J. Antimicrob. Chemother. 72 , 3016–3024 (2017).
-
Ching, C. y Zaman, MH. Desarrollo y selección de resistencia multifármaco de bajo nivel en un rango extendido de concentraciones subinhibitorias de ciprofloxacino en Escherichia coli. Sci. Rep. 10 , 8754 (2020).
-
Gerken, H. y Misra, R. Evidencia genética de interacciones funcionales entre las proteínas TolC y AcrA de una importante bomba de eflujo de antibióticos de Escherichia coli. Mol. Microbiol. 54 , 620–631 (2004).
-
Pérez, A. et al. Efecto de los activadores transcripcionales SoxS, RobA y RamA en la expresión de la bomba de eflujo multifármaco AcrAB-TolC en Enterobacter cloacae. Antimicrobiano. Agentes Chemother. 56 , 6256–6266 (2012).
-
Aly, SA, Boothe, DM y Suh, S.-J. Una nueva mutación de sustitución de alanina a serina en SoxS induce la sobreexpresión de bombas de eflujo y contribuye a la resistencia a múltiples fármacos en aislados clínicos de Escherichia coli. J. Antimicrob. Chemother. 70 , 2228–2233 (2015).
-
Moon, DC et al. La aparición de una nueva mutación y su acumulación en el gen de la topoisomerasa IV confiere altos niveles de resistencia a las fluoroquinolonas en aislados de Escherichia coli. Int. J. Antimicrobiano, agentes 35 , 76–79 (2010).
-
Johnning, A. et al. Las mutaciones de resistencia en gyrA y parC son comunes en comunidades de Escherichia de ambientes acuáticos contaminados y no contaminados con fluoroquinolonas. Front. Microbiol. 6 , 1355 (2015).
-
Hawkey, PM Mecanismos de acción de las quinolonas y respuesta microbiana. J. Antimicrob. Chemother. 51 , 29–35 (2003).
-
Hall, RJ et al. Los fármacos no antibióticos son tóxicos contra Escherichia coli sin que se desarrolle resistencia cruzada a los antibióticos. npj Antimicrob. Resist. 2 , 11 (2024).
-
Li, L. et al. Análisis de los mecanismos sinérgicos de la tetraciclina y la sertralina, inhibidor selectivo de la recaptación de serotonina, en una cepa de Escherichia coli resistente a la tetraciclina. J. Antibiot. 70 , 944–953 (2017).
-
Theodorakis, N. et al. Resistencia a los antibióticos en ancianos: mecanismos, factores de riesgo y soluciones. Microorganisms 12 , 1978 (2024).
-
Tawam, D. et al. Asociación positiva entre los inhibidores de la bomba de protones y la infección por Clostridium difficile. Innov. Pharm. 12, https://doi.org/10.24926/iip.v24912i24921.23439 (2021).
-
Gordon, D. et al. Incidencia de infección por Clostridium difficile en pacientes que reciben antibióticos de alto riesgo con o sin inhibidores de la bomba de protones. J. Hosp. Infect. 92 , 173–177 (2016).
-
Chen, EY et al. Complejidad del régimen de medicación en ocho residencias geriátricas australianas: impacto de la edad, la duración de la estancia, la comorbilidad, la fragilidad y la dependencia en las actividades de la vida diaria. Clin. Interven. Aging 14 , 1783-1795 (2019).
-
Grenier, F., Matteau, D., Baby, V., y Rodrigue, S. Secuencia genómica completa de Escherichia coli BW25113. Anuncios del genoma , 2, https://doi.org/10.1128/genomea.01038-01014 (2014).
-
Datsenko, KA y Wanner, BL. Inactivación en un solo paso de genes cromosómicos en Escherichia coli K-12 mediante productos de PCR. Proc. Natl. Acad. Sci. 97 , 6640–6645 (2000).
-
Centro Colaborador de la OMS para la Metodología de Estadísticas de Medicamentos, Directrices para la clasificación ATC y la asignación de DDD. (1996).
-
Eucast, D. Determinación de las concentraciones mínimas inhibitorias (CMI) de agentes antibacterianos mediante dilución en agar. Clin. Microbiol. Infect. 6 , 509–515 (2000).
-
Jin, M. et al. El antidepresivo fluoxetina induce resistencia a múltiples antibióticos en Escherichia coli mediante mutagénesis mediada por ROS. Environ. Int. 120 , 421–430 (2018).
-
Pisoni, LA et al. Enfoque basado en la estructura y el ligando para la identificación de inhibidores de AcrAB-TolC en Escherichia coli. ACS Infect. Dis. 9 , 2504–2522 (2023).
-
Blaikie, JM et al. El análisis resistómico del complejo Klebsiella pneumoniae de centros residenciales de atención a la tercera edad demuestra la propagación clonal intracentro de aislamientos multirresistentes. Microorganisms 12 , 751 (2024).
-
Ohene-Agyei, T., Mowla, R., Rahman, T. y Venter, H. Los fitoquímicos aumentan la actividad antibacteriana de los antibióticos al actuar sobre una bomba de eflujo de fármacos. Microbiologyopen 3 , 885–896 (2014).
-
Quijada, NM, Rodríguez-Lázaro, D., Eiros, JM & Hernández, M. TORMES: un proceso automatizado para el análisis del genoma bacteriano completo. Bioinformática 35 , 4207–4212 (2019).
-
Barrick, JE et al. Identificación de la variación estructural en genomas microbianos haploides a partir de datos de resecuenciación de lectura corta mediante breseq. BMC Genomics 15 , 1–17 (2014).
-
Sievers, F. y Higgins, DG Clustal Omega para realizar alineamientos precisos de numerosas secuencias de proteínas. Protein Sci. 27 , 135–145 (2018).
-
Robert, X. y Gouet, P. Descifrando características clave en las estructuras proteicas con el nuevo servidor ENDscript. Nucleic Acids Res. 42 , W320–W324 (2014).
Expresiones de gratitud
Agradecemos al proveedor de atención a personas mayores y al Sr. Barry Lowe por la recolección de muestras y las listas de medicamentos de uso común en los centros de atención a personas mayores con discapacidad (RACF), así como a la profesora adjunta Janet Slugget (UniSA) por su asesoramiento sobre la polifarmacia y el uso de medicamentos en los RACF de Australia. También agradecemos al Dr. Lex Leong (Patología de SA) por la secuenciación completa del genoma. La financiación del proyecto para este estudio proviene de Medical Research Future (MRFF, GN1152556) para HV y JT. CH ha recibido una beca RTPi.
Declaraciones éticas
Intereses en competencia
Los autores declaran no tener intereses en conflicto.
Información adicional
Nota del editor: Springer Nature se mantiene neutral con respecto a los reclamos jurisdiccionales en los mapas publicados y las afiliaciones institucionales.